- 고객지원
- 업무안내│제품문의
- 전화번호│02-2081-2510
- Email │support@takara.co.kr
- 업무시간안내
- [ 평 일 ] 09 : 00 ~ 18 : 00 │ [ 점심시간 ] 12 : 00 ~ 13 : 00
- 토·일요일, 공휴일은 휴무입니다.
[적용] 발톱개구리 초기배아에 대한 ChIP-Seq


발톱개구리 초기배아에 대한 ChIP-Seq
Data kindly provided by: 이화학연구소 (RIKEN)
◆ Introduction
ChIP-Seq은 염색질 면역침강 (ChIP)을 이용하여 목적 단백질이 결합되어 있는 염색체 영역을 농축한 뒤, 차세대 시퀀싱 (Next Generation Sequencing)으로 서열을 분석하는 기법이다. 동물의 발생 과정에서 세포의 운명을 결정하는 전사인자 단백질과 DNA의 상호작용이 세포 발생을 어떻게 제어하는지 규명하는 데 있어, 동물 배아를 이용한 ChIP-Seq 분석은 매우 중요하다. 하지만 동물 배아의 일부 세포에서만 발현되는 발생조절인자 (전사인자)에 대하여 ChIP-Seq 분석을 진행하는 경우, 적은 양의 세포에서 농축된 극소량의 ChIP DNA를 이용하여 NGS 분석을 진행해야 한다. 본 연구실에서 이용한 발톱개구리 (Xenopus sp.)는 한꺼번에 배아를 획득 가능하며, 초기 발생 유전자의 발현 제어를 분석하는데 적합한 모델로써 본 연구에서는 소량의 발톱개구리 배아에서 농축한 미량의 ChIP DNA를 ThruPLEX® DNA-Seq Kit을 이용하여 ChIP-Seq 분석을 진행하였다.
◆ 사용제품
ThruPLEX® DNA-Seq Kit (Code 400674)
◆ Workflow
① 배아 고정: 발톱개구리의 낭배 (gastrula) 약 250개를 1% 포르말린으로 고정 후, -80℃에서 냉동 보존
② 세포 용해와 핵 농축:
계면활성제가 포함된 Lysis Buffer로 배아를 균질화 (homogenize) 후, 핵 농축
③ Chromatin fragmentation:
초음파세포파쇄장치 Picoruptor (Diagenode)를 이용하여 크로마틴 단편화
④ 면역침강 (Immuno-precipitation):
Anti-Otx2 항체를 미리 결합시킨 Protein A Magnetic Beads와 상기의 단편화된 크로마틴을 혼합하고 4℃에서 overnight 교반 후 RIPA Buffer로 washing
⑤ Cross-link reversal:
1% SDS를 포함한 용출 buffer에서 65℃, overnight incubation 후, cross-link reversal beads 하에서 RNase A 및 Proteinase K를 처리하여 cross-link reversal
⑥ DNA 정제 (DNA Purification): MinElute PCR Purification Kit (Qiagen)으로 ChIP DNA 정제
⑦ ChIP DNA Quality Control (QC):
형광광도계 (Qubit dsDNA HS Kit)을 이용하여 ChIP DNA 정량하고, qPCR로 게놈영역 농축 확인
⑧ NGS Library Preparation:
ThruPLEX® DNA-Seq Kit을 이용하여 10 ㎕의 ChIP DNA로부터 NGS Library Preparation
⑨ Sequencing: Illumina® NestSeq550, 75 bp paired-end
⑩ Data analysis: BWA 소프트웨어를 이용하여 발톱개구리 게놈 (v9)에 mapping, IGV visualization
◆ NGS Mapping 결과
샘플 |
Total reads |
Mapped reads (%) |
Input |
74,426,066 |
70,219,181 (94.35%) |
Otx2 ChIP |
82,794,598 |
75,710,767 (91.44%) |
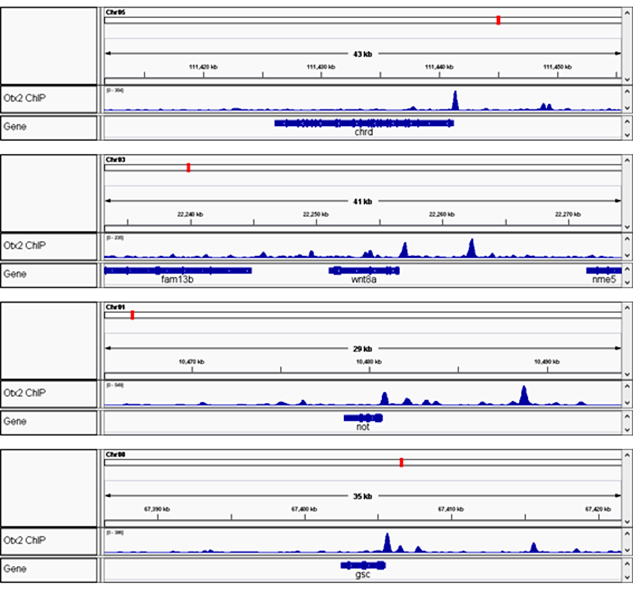
◆ Otx2 ChIP-seq 결과
250개의 발톱개구리 배아로부터 획득한 1 ng 정도의 ChIP DNA에서 충분한 양의 NGS Library를 제작하여 분석할 수 있었다 (PCR Cycle = 12). 또한 Mapping data에서 약 2,000개의 발톱개구리 배아로부터 수행한 ChIP-Seq 분석 결과와 동일함을 알 수 있다 (Yasuoka et al., Nature Communications 2014 5 : 4322)
◆ 향후 계획
ThruPLEX® DNA-Seq Kit를 이용하면 극소량의 DNA에서 NGS Library 제작이 가능하기 때문에 동일한 전사인자에 대해 반복적인 ChIP-Seq 분석을 시행하여, 전사인자가 어느 정도의 차이 (variation)을 가지고 결합하는지에 대해 향후 규명할 예정이다 (http://constrained-evo.org/proposed_projects.html).
표적으로 하는 전사인자와 사용하는 항체에 따라 다를 수 있지만, 본 제품을 이용하면 ChIP에 필요한 배아의 수를 50 ~ 100개 정도로 줄일 수 있어, 보다 효율적으로 연구를 진행할 수 있을 것으로 기대된다.
◆ 제품 사용을 검토 중인 분께 사용자의 한마디
ThruPLEX® DNA-Seq Kit는 형광 광도계에서도 측정이 불가능할 정도로 극소량인 DNA에서 NGS Library 제작이 가능하므로, 아주 적은 양의 세포에 대해 ChIP-Seq 분석을 실시할 경우 추천합니다. 또한 실험의 과정이 매우 간편하고 제품 내의 각 구성품들도 각기 다른 라벨로 구분이 되어 있어 매우 쉽게 NGS Library를 제작할 수 있으므로, NGS Library 제작에 익숙하지 않은 사람에게도 매우 적합합니다.
ThruPLEX® DNA-Seq Kit는 형광 광도계에서도 측정이 불가능할 정도로 극소량인 DNA에서 NGS Library 제작이 가능하므로, 아주 적은 양의 세포에 대해 ChIP-Seq 분석을 실시할 경우 추천합니다. 또한 실험의 과정이 매우 간편하고 제품 내의 각 구성품들도 각기 다른 라벨로 구분이 되어 있어 매우 쉽게 NGS Library를 제작할 수 있으므로, NGS Library 제작에 익숙하지 않은 사람에게도 매우 적합합니다.