Seq-ing the small: metagenomics and metatranscriptomics
미생물은 다양하고 복잡하며, 이를 분석하기 위해서는 전문적인 접근 방식이 요구된다. 최근 들어, 대량 샘플을 높은 민감도로 sequencing하는 방식이 개발되면서, 매우 다양한 미생물 집단의 DNA-seq (metagenome)과 RNA-seq (metatranscriptomics)이 가능해졌다. 다카라바이오가 제공하는
ThruPLEX® DNA-Seq Kit와
SMARTer® Stranded RNA-Seq Kit를 이용해 미생물을 분석한 연구 사례는 아래에서 확인할 수 있다.
Metagenomics and the growing (antibiotic) resistance
건강은 환경과 밀접하게 관계되어 있으며, 특히 미생물이 빠질 수 없다. 이를 설명하는 단적인 예로 인간 배설물 (human waste)을 들어볼 수 있는데, 이로 인해 우리 주변에 존재하는 미생물이 항생제 저항성을 획득할 수 있다.
노르웨이 환경청에서 작성한 한 보고서에서 연구자들은 환경적 요인으로 인한 항생제 내성균과 항생제 내성 유전자 간의 상관 관계를 확인하고자 했다. 이를 위해 노르웨이 전역의 6 종의 오수 처리 시설을 분석하고, 이의 상관 관계를 모니터링 했다.
높은 수율로 DNA를 추출하는 것은 과정이 복잡하고 어렵기 때문에, 각 시설에서 10개의 정제되지 않은 오수 샘플을 채취하여 DNA를 추출하였다. 추출된 DNA는
ThruPLEX® DNA-Seq Kit를 이용해 Metagenomic library로 제작하였다. 시퀀싱을 진행한 결과, 110개의 항생제 내성 유전자가 일치하는 것을 확인했으며, 그 중 99개는
mex-family efflux pump를 이용하는 것으로 확인되었다. 이를 통해 연구자들은 확인한 유전자 중 일부가 항생제 내성균과 관련되어 있으며,
mex-family efflux pump로 인해 미생물의 숙주 내 잔존율이 높아지고, 발병을 촉진할 수 있음을 입증했다 (Piddock 2006).
⇒ Why is this important?
Metagenomics는 미생물 유전체를 대량으로 분석하는 것을 가능케 하며, 이 연구는 항생제 내성 유전자에 대한 환경 샘플을 스크리닝하는 과정에서 metagenomics를 활용했다. 이 보고서에서는 환경이 항생제 내성을 전달하는 저장소 역할을 할 수 있기에, 이를 모니터링 하는 것이 중요함을 강조하고 있다. 하지만 분석하고자 하는 샘플의 복잡성, 적은 샘플 양, 낮은 처리량을 가지는 실험 과정으로 인해 분석 과정에 어려움이 있을 수 있다. 해당 보고서에서 활용한 것처럼, 다카라바이오의
ThruPLEX® DNA-Seq Kit를 사용하면, 단일 tube 내에서 진행되는 실험방식으로 편리하게 대량의 metagenomic library를 제작할 수 있다.
Metatranscriptomics: turning fibers into fuel
흰개미는 복합 탄수화물을 소화할 수 있는 장내 미생물이 있어, 나무를 먹는 것으로 유명하다. 이는 흰개미의 장내에서 식물성 건조 물질인 lignocellulosic biomass의 처리를 위한 새로운 효소를 발견할 가능성이 있음을 시사한다. 하지만, 안타깝게도 흰개미의 장에서부터 sequencing library를 제작하는 것에 기술적인 한계가 있었다.
이 기술적 한계를 극복하기 위해, University of California, Irvine의 그룹은 다카라바이오의 RNA-seq 기술을 이용해, 흰개미의 장내 metatranscriptomic 분석을 진행하는 방법을 개발했다 (Marynowska
et al. 2017). 연구원들은
SMARTer® Stranded RNA-Seq Kit를 이용해 4종의 흰개미 내장에 대한 library를 제작하였다.
Illumina
® MiSeq
® 플랫폼에서 이 library를 분석한 결과, 전체 mRNA 중 최대 14%가 탄수화물 대사 및 수송 유전자인 것으로 확인되었다. 주목할 만한 점은 이 연구를 통해 116개의 계열에 해당하는 1,643개의 효소를 식별했다는 점이다 (
그림 1).
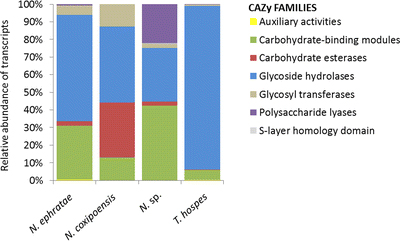 그림 1. 흰개미의 탄수화물 관련 효소의 식별과 분류
그림 1. 흰개미의 탄수화물 관련 효소의 식별과 분류
Metatranscriptomic analysis of the gut microbiome from each of four termite species reveals a unique "fingerprint" of carbohydrate-active enzymes dispersed across several different classes.
⇒ Why is this important?
Lignocellulosic biomass는 바이오 연료 생산을 위한 가장 좋은 원료지만, 현재 사용되고 있는 처리 방식은 많은 비용이 요구된다. 바이오 연료의 고효율 촉매 작용을 가능하게 하는 효소로 처리방식을 대체한다면, 보다 저렴하고 재생 가능한 에너지원을 얻을 수 있다. 새로운 효소를 확인하기 위한 흰개미 장내 미생물 군집 분석은 군집의 복잡성과 초기 샘플의 양이 적어 그간 분석이 어려웠으나, 다카라바이오의
SMARTer® Stranded RNA-Seq Kit를 이용하면. 이를 해결할 수 있음이 확인되었다.
The future of the small
최근 DNA, RNA sequencing 기술이 대량으로 적용될 수 있도록 발전하면서, 기존에는 불가능했던 metagenomics, metatranscriptomics 분석이 가능해 졌으나 여전히 분석 과정에서 어려움이 존재한다. 샘플이 복잡할수록, 샘플 양이 적을수록, 샘플 수가 많을수록 분석 과정에 어려움이 발생할 수 있지만, 다카라바이오는 이를 극복하고 극도로 적은 양의 샘플로부터 원하는 데이터를 얻을 수 있도록
ThruPLEX® DNA-Seq Kit와
SMARTer® Stranded RNA-Seq Kit 기술을 통해 솔루션을 제공하고 있다.
[References]
- Marynowska, M.
et al. Optimization of a metatranscriptomic approach to study the lignocellulolytic potential of the higher termite gut microbiome. BMC Genomics 18, 681 (2017).
- Piddock, L. J. V. Multidrug-resistance efflux pumps - not just for resistance. Nat. Rev. Microbiol. 4, 629-636 (2006).
[원문] Seq-ing the small: metagenomics and metatranscriptomics





