- 고객지원
- 업무안내│제품문의
- 전화번호│02-2081-2510
- Email │support@takara.co.kr
- 업무시간안내
- [ 평 일 ] 09 : 00 ~ 18 : 00 │ [ 점심시간 ] 12 : 00 ~ 13 : 00
- 토·일요일, 공휴일은 휴무입니다.
[Cancer Research] Cancer single cell analysis

Single cancer cell analysis
종양세포의 주요한 유전적 변화는 게놈의 일부 혹은 유전자가 결실, 전위, 증폭되는 구조적 이유일수도 있고, 돌연변이와 같은 DNA 서열 자체의 영향일 수 있다. 이러한 돌연변이를 포함하는 개체가 많아지면서 암이 진행될 수 있으며, 종양 DNA의 특성으로 수많은 악성 변이가 나타날 수 있다. 최근 NGS 기술이 발전되며, 단일 세포 수준에서 종양세포의 생성과 계통 발생, 이질성 (heterogeneity), 항암 치료의 영향 모니터링 등 DNA 돌연변이를 체계적으로 분석할 수 있게 되었다 (Van Loo and Voet, 2014). 종양세포에서 나타나는 복잡성 (Complexity)과 이질성 (Heterogeneity)은 종양세포 (cancer cell), 지속성 암세포 (Cancer persister cells), 순환종양세포 (Circulating Tumor Cell; CTC)를 단일세포 수준에서 해석함으로써 분석 가능하다.
지속성 암세포나 CTC와 같은 얻기 힘든 샘플에서 단일종양세포를 연구하기 위해서는, 단일종양세포 내 RNA 전사체 변이를 정확하게 검출하여 정량화 할 수 있는 아주 민감하고 재현성 높은 분석 방법이 필요하다. 이를 통해 종양세포의 복잡성을 확인하고, 궁극적으로 맞춤형 항암 치료제의 개발에 활용할 수 있다. 다카라바이오에서는 독자적인 SMARTer® 기술을 이용하여 단일세포 수준의 염기서열 분석을 위한 제품을 제공한다.
Highlighted products - Single-cell genome sequencing
PicoPLEX® 기술을 이용한 PicoPLEX® Gold Single Cell DNA-seq Kit는 단일 세포의 whole genome amplification (WGA)를 위한 제품으로, quasi-random priming을 이용해 Illumina® 플랫폼에서 sequencing 가능한 library를 재현성 높게 제작할 수 있다. 실제로, PicoPLEX® 기술은 유전자복제변이 (copy number variation ,CNV), 염색체 이수성 (Chromosomal aneuploidies), 단일 염기 치환 (single nucleotide variation, SNV) 등을 포함하여 종양 연구에서 사용되는 게놈의 다양한 분석에 적용할 수 있다 (그림 1). 본 기술을 이용하여 FFPE 조직, CTC에서 추출한 단일 세포의 게놈이나 CNV의 분석 시 높은 성능을 확인한 논문이 다수 출판되고 있을 뿐만 아니라, 최근에는 단일 세포의 Genome & Transcriptome-seq (G&T-seq)의 필수 구성 요소로 꼽히고 있다 (Babayan et al. 2017, Cayrefourcq et al. 2015, Lieselot et al. 2017, Macaulay et al. 2015, Morrow et al. 2016, Premasekharan et al. 2016, Williamson et al. 2016).
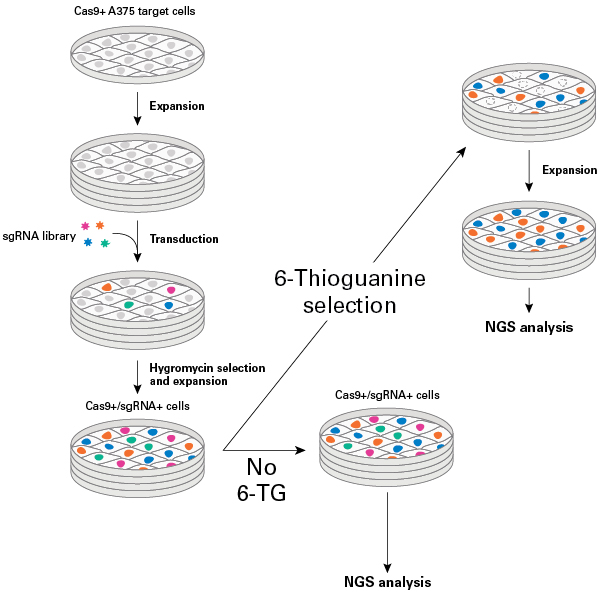
그림 1. PicoPLEX® Gold Single Cell DNA-seq Kit을 이용한 두 개의 세포에서 CNV 검출
Log2 ratio of the total number of reads in 50-kb bins from single NCI-H929 cells, shown as one cell in Panel A and a second cell in Panel B. Red bars represent copy-number gains while blue bars represent losses. The top row of the graphs in each panel depicts the control bulk sample sequenced to a depth of 90 million read pairs. The highly reproducible coverage of the PicoPLEX® Gold kit enables the accurate detection of structural variants as small as 100 kb, even at shallow sequencing depths (2.5-8.5 million read pairs).
다카라바이오는 SMART-Seq® 기술을 이용해 full-length mRNA 서열을 분석할 수 있는 NGS 제품을 제공함으로써, 단일 세포 mRNA-seq 분석 연구에 앞장서고 있다. 4세대인 SMART-Seq® v4 PLUS Kit와 이를 기반으로 대량 분석에 최적화되어 있는 SMART-Seq® HT Kit를 이용하면 단일세포 수준의 극소량 샘플과 RNA로부터 가장 높은 민감도로 mRNA-seq 분석을 할 수 있다. 이 제품들은 oligo(dT) primer와 SMART (Switching Mechanism at 5' end of RNA Template) 기술을 이용하여 full-length mRNA를 편향성 없이 분석할 수 있으며, intact 상태의 세포를 바로 샘플로 적용하였을 때도 고품질로 cDNA를 합성할 수 있다. 본 SMART-Seq® v4 기술은 단일 세포를 대량으로 분석하기 위한 자동화 시스템인 Apollo™ Library Prep System과 ICELL8® Single-Cell System에 적용할 수 있다. 많은 논문에서 이 SMART-Seq® 방법을 이용한 단일 세포의 RNA-seq을 채택하여 종양 세포나 종양 침윤 면역 세포에서의 게놈, 전사체를 sequencing 하여 분석하거나, 암 줄기세포의 이질성을 분석하고, 약제 내성을 가지는 종양 세포를 식별하는 등 다양한 종양 세포 연구를 진행하였다 (Chung et al. 2017, Chiu et al. 2018, Han et al. 2018, Kim et al. 2015, and Zheng et al. 2018).
SMART-Seq Stranded Kit는 SMARTer Stranded Total RNA-Seq Kit v2 - Pico Input Mammalian Kit의 독특한 특징과 SMART-Seq® v4 기술을 결합하여 단일 세포에서 Illumina®에 호환 가능한 stranded sequencing library를 제작할 수 있도록 개발되었다. 이는 종양 FFPE 조직과 같이 매우 품질이 낮은 pg 수준의 total RNA로부터 library를 제작하는데 최적의 솔루션이다.
[원문] Single cancer cell analysis
[참고문헌]
- Babayan, A. et al. Comparative study of whole genome amplification and next-generation sequencing performance of single cancer cells. Oncotarget 8, 56066-56080 (2017).
- Cayrefourcq, L. et al. Establishment and Characterization of a Cell Line from Human Circulating Colon Cancer Cells. Cancer Res. 75, 892-901 (2015).
- Chiu, H. S. et al. Pan-Cancer Analysis of lncRNA Regulation Supports Their Targeting of Cancer Genes in Each Tumor Context. Cell Rep. 23, 297-312 (2018).
- Chung, W. et al. Single-cell RNA-seq enables comprehensive tumour and immune cell profiling in primary breast cancer. Nature Comm. 8, 15081 (2017).
- Han, K. Y. et al. SIDR: simultaneous isolation and parallel sequencing of genomic DNA and total RNA from single cells. Genome Res. 28, 75-87 (2018).
- Kim, K. T. et al. Single-cell mRNA sequencing identifies subclonal heterogeneity in anti-cancer drug responses of lung adenocarcinoma cells. Genome Biol. 16, 127 (2015).
- Lieselot, D. et al. Performance of four modern whole genome amplification methods for copy number variant detection in single cells. Sci. Rep. 7, 3422 (2017).
- Macaulay, I. C. et al. G&T-seq: parallel sequencing of single-cell genomes and transcriptomes. Nature Methods 12, 519-522 (2015).
- Morrow, C. J. et al. Tumourigenic non-small-cell lung cancer mesenchymal circulating tumour cells: a clinical case study. Ann. Oncol. 27, 1155-1160 (2016).
- Premasekharan, G. et al. An improved CTC isolation scheme for pairing with downstream genomics: Demonstrating clinical utility in metastatic prostate, lung and pancreatic cancer. Cancer Letters 380, 144-152 (2016).
- Van Loo, P. & Voet, T. Single-cell analysis of cancer genomes. Current Opinion in Genetics & Development 24, 82-91 (2014).
- Williamson, S. C. et al. Vasculogenic mimicry in small cell lung cancer. Nature Comm. 7, 13322 (2016).
- Zheng, H. et al. Single-cell analysis reveals cancer stem cell heterogeneity in hepatocellular carcinoma. Hepatology doi: 10.1002/hep.29778 (2018).
- Zhu, S. et al. Advances in single-cell RNA sequencing and its applications in cancer research. Oncotarget 16, 53763-53779 (2017).
종양세포의 주요한 유전적 변화는 게놈의 일부 혹은 유전자가 결실, 전위, 증폭되는 구조적 이유일수도 있고, 돌연변이와 같은 DNA 서열 자체의 영향일 수 있다. 이러한 돌연변이를 포함하는 개체가 많아지면서 암이 진행될 수 있으며, 종양 DNA의 특성으로 수많은 악성 변이가 나타날 수 있다. 최근 NGS 기술이 발전되며, 단일 세포 수준에서 종양세포의 생성과 계통 발생, 이질성 (heterogeneity), 항암 치료의 영향 모니터링 등 DNA 돌연변이를 체계적으로 분석할 수 있게 되었다 (Van Loo and Voet, 2014). 종양세포에서 나타나는 복잡성 (Complexity)과 이질성 (Heterogeneity)은 종양세포 (cancer cell), 지속성 암세포 (Cancer persister cells), 순환종양세포 (Circulating Tumor Cell; CTC)를 단일세포 수준에서 해석함으로써 분석 가능하다.
지속성 암세포나 CTC와 같은 얻기 힘든 샘플에서 단일종양세포를 연구하기 위해서는, 단일종양세포 내 RNA 전사체 변이를 정확하게 검출하여 정량화 할 수 있는 아주 민감하고 재현성 높은 분석 방법이 필요하다. 이를 통해 종양세포의 복잡성을 확인하고, 궁극적으로 맞춤형 항암 치료제의 개발에 활용할 수 있다. 다카라바이오에서는 독자적인 SMARTer® 기술을 이용하여 단일세포 수준의 염기서열 분석을 위한 제품을 제공한다.
Highlighted products - Single-cell genome sequencing
PicoPLEX® 기술을 이용한 PicoPLEX® Gold Single Cell DNA-seq Kit는 단일 세포의 whole genome amplification (WGA)를 위한 제품으로, quasi-random priming을 이용해 Illumina® 플랫폼에서 sequencing 가능한 library를 재현성 높게 제작할 수 있다. 실제로, PicoPLEX® 기술은 유전자복제변이 (copy number variation ,CNV), 염색체 이수성 (Chromosomal aneuploidies), 단일 염기 치환 (single nucleotide variation, SNV) 등을 포함하여 종양 연구에서 사용되는 게놈의 다양한 분석에 적용할 수 있다 (그림 1). 본 기술을 이용하여 FFPE 조직, CTC에서 추출한 단일 세포의 게놈이나 CNV의 분석 시 높은 성능을 확인한 논문이 다수 출판되고 있을 뿐만 아니라, 최근에는 단일 세포의 Genome & Transcriptome-seq (G&T-seq)의 필수 구성 요소로 꼽히고 있다 (Babayan et al. 2017, Cayrefourcq et al. 2015, Lieselot et al. 2017, Macaulay et al. 2015, Morrow et al. 2016, Premasekharan et al. 2016, Williamson et al. 2016).

그림 1. PicoPLEX® Gold Single Cell DNA-seq Kit을 이용한 두 개의 세포에서 CNV 검출
Log2 ratio of the total number of reads in 50-kb bins from single NCI-H929 cells, shown as one cell in Panel A and a second cell in Panel B. Red bars represent copy-number gains while blue bars represent losses. The top row of the graphs in each panel depicts the control bulk sample sequenced to a depth of 90 million read pairs. The highly reproducible coverage of the PicoPLEX® Gold kit enables the accurate detection of structural variants as small as 100 kb, even at shallow sequencing depths (2.5-8.5 million read pairs).
다카라바이오는 SMART-Seq® 기술을 이용해 full-length mRNA 서열을 분석할 수 있는 NGS 제품을 제공함으로써, 단일 세포 mRNA-seq 분석 연구에 앞장서고 있다. 4세대인 SMART-Seq® v4 PLUS Kit와 이를 기반으로 대량 분석에 최적화되어 있는 SMART-Seq® HT Kit를 이용하면 단일세포 수준의 극소량 샘플과 RNA로부터 가장 높은 민감도로 mRNA-seq 분석을 할 수 있다. 이 제품들은 oligo(dT) primer와 SMART (Switching Mechanism at 5' end of RNA Template) 기술을 이용하여 full-length mRNA를 편향성 없이 분석할 수 있으며, intact 상태의 세포를 바로 샘플로 적용하였을 때도 고품질로 cDNA를 합성할 수 있다. 본 SMART-Seq® v4 기술은 단일 세포를 대량으로 분석하기 위한 자동화 시스템인 Apollo™ Library Prep System과 ICELL8® Single-Cell System에 적용할 수 있다. 많은 논문에서 이 SMART-Seq® 방법을 이용한 단일 세포의 RNA-seq을 채택하여 종양 세포나 종양 침윤 면역 세포에서의 게놈, 전사체를 sequencing 하여 분석하거나, 암 줄기세포의 이질성을 분석하고, 약제 내성을 가지는 종양 세포를 식별하는 등 다양한 종양 세포 연구를 진행하였다 (Chung et al. 2017, Chiu et al. 2018, Han et al. 2018, Kim et al. 2015, and Zheng et al. 2018).
SMART-Seq Stranded Kit는 SMARTer Stranded Total RNA-Seq Kit v2 - Pico Input Mammalian Kit의 독특한 특징과 SMART-Seq® v4 기술을 결합하여 단일 세포에서 Illumina®에 호환 가능한 stranded sequencing library를 제작할 수 있도록 개발되었다. 이는 종양 FFPE 조직과 같이 매우 품질이 낮은 pg 수준의 total RNA로부터 library를 제작하는데 최적의 솔루션이다.
Code |
제품명 |
용량 |
R300669 |
24 회 |
|
R400752 |
48 회 |
|
R400748 |
48 회 |
|
640170 |
96 회 |
|
640189 |
1 system |
|
634442 |
12 회 |
|
634411 |
SMARTer® Stranded Total RNA-Seq Kit v2 - Pico Input Mammalian |
12 회 |
[원문] Single cancer cell analysis
[참고문헌]
- Babayan, A. et al. Comparative study of whole genome amplification and next-generation sequencing performance of single cancer cells. Oncotarget 8, 56066-56080 (2017).
- Cayrefourcq, L. et al. Establishment and Characterization of a Cell Line from Human Circulating Colon Cancer Cells. Cancer Res. 75, 892-901 (2015).
- Chiu, H. S. et al. Pan-Cancer Analysis of lncRNA Regulation Supports Their Targeting of Cancer Genes in Each Tumor Context. Cell Rep. 23, 297-312 (2018).
- Chung, W. et al. Single-cell RNA-seq enables comprehensive tumour and immune cell profiling in primary breast cancer. Nature Comm. 8, 15081 (2017).
- Han, K. Y. et al. SIDR: simultaneous isolation and parallel sequencing of genomic DNA and total RNA from single cells. Genome Res. 28, 75-87 (2018).
- Kim, K. T. et al. Single-cell mRNA sequencing identifies subclonal heterogeneity in anti-cancer drug responses of lung adenocarcinoma cells. Genome Biol. 16, 127 (2015).
- Lieselot, D. et al. Performance of four modern whole genome amplification methods for copy number variant detection in single cells. Sci. Rep. 7, 3422 (2017).
- Macaulay, I. C. et al. G&T-seq: parallel sequencing of single-cell genomes and transcriptomes. Nature Methods 12, 519-522 (2015).
- Morrow, C. J. et al. Tumourigenic non-small-cell lung cancer mesenchymal circulating tumour cells: a clinical case study. Ann. Oncol. 27, 1155-1160 (2016).
- Premasekharan, G. et al. An improved CTC isolation scheme for pairing with downstream genomics: Demonstrating clinical utility in metastatic prostate, lung and pancreatic cancer. Cancer Letters 380, 144-152 (2016).
- Van Loo, P. & Voet, T. Single-cell analysis of cancer genomes. Current Opinion in Genetics & Development 24, 82-91 (2014).
- Williamson, S. C. et al. Vasculogenic mimicry in small cell lung cancer. Nature Comm. 7, 13322 (2016).
- Zheng, H. et al. Single-cell analysis reveals cancer stem cell heterogeneity in hepatocellular carcinoma. Hepatology doi: 10.1002/hep.29778 (2018).
- Zhu, S. et al. Advances in single-cell RNA sequencing and its applications in cancer research. Oncotarget 16, 53763-53779 (2017).