- 고객지원
- 업무안내│제품문의
- 전화번호│02-2081-2510
- Email │support@takara.co.kr
- 대전지사
- 업무안내│대전/충청지역 주문, 제품문의
- 전화번호│042-828-6525
- Email │tkbd@takara.co.kr
- 업무시간안내
- [ 평 일 ] 09 : 00 ~ 18 : 00 │ [ 점심시간 ] 12 : 00 ~ 13 : 00
- 토·일요일, 공휴일은 휴무입니다.
[Cancer Research] Cancer biomarker discovery

EV를 이용한 total RNA-seq
The unrealized potential of cell-free RNA
RNA profiling은 사람 체액으로부터 biomarker를 확인하기 위해 사용되는 강력한 도구이다. 액체 생검 분석을 위해 Circulating DNA가 가장 보편적으로 사용되고 있지만, 혈장, 소변, extracellular vesicles (EV) 로부터 추출한 extracellular RNA 또한 질병 상태에 대한 유용한 정보를 제공할 수 있다. 하지만, 체액을 이용해 RNA를 sequencing하는 것은 샘플 양이 적거나, 정량하기 어려울 정도로 RNA가 손상되는 등으로 기술적인 한계가 있었다.
게다가, 최근의 Cell-free 샘플의 total RNA sequencing을 이용한 전사체 분석법은 짧은 fragment, 발현량이 적은 유전자, rRNA의 오염 등으로 인한 분석 한계가 있었다. 이에 대한 대안으로, Ghent 대학의 유전체 연구소와 Biogazelle, Zwijnaarde (Belgium) 연구원들은 다양한 체액과 EV로부터 total RNA를 정확하고, 민감도 있게 정량하기 위해, SMARTer® Stranded Total RNA-Seq Kit - Pico Input Mammalian (Pico v2)로 strand-specific total RNA library를 제작하여 평가하였다.
[Putting Pico v2 to the test] Read distribution and sample type
두 공여자의 platelet-rich plasma in EDTA tube (ePRP), platelet-free plasma in EDTA tube (ePFP)로부터 RNA-seq을 진행하였다. ePRP에서는 75% 이상의 unique reads가 mitochondrial RNA (mtRNA)로부터 유래로 확인된 반면, ePFP는 mtRNA 유래의 reads가 3배 적었고, 많은 reads가 nuclear DNA에 mapping 되었다. 혈장 내 혈소판 유무와 관계없이 unmapped reads와 multi-mapped reads의 수는 비슷하게 확인되었지만, exonic, intronic, intergenic reads 분포는 혈장 내 혈소판 유무에 따라 다르게 나타났다 (그림 1).
함께, 본 논문의 저자는 유방암 세포의 배양 배지 (conditioned medium, CM), 건강한 세 번째 공여자의 platelet-free plasma in citrate tube (cPFP), 전립선 암 환자의 소변 샘플 그리고, 각 샘플 내 EV를 이용하여 염기서열을 분석하였다. cPFP 유래 EV는 7.69%의 unique reads만 확인된 반면 CM 유래 EV에는 90.2%의 unique reads가 확인되었을 만큼 각각의 샘플과 EV에서 다양한 mapping rate를 보였다 (그림 1, Panal A). 거의 대부분의 샘플에서 확인된 Unique reads는 주로 nuclear DNA에 mapping되었으며, 건강한 공여자의 cPFP는 25.8%의 mtRNA를 포함하고 있음을 확인하였다 (그림 2, Panal B). Intron과 intergenic reads를 다수 포함하고 있는 cPFP 유래 EV를 제외하고는, nuclear DNA에 mapping되는 대부분의 reads는 exon 유래임이 확인 되었다 (그림 1, Panal C).
SMARTer® Stranded Total RNA-Seq Kit - Pico Input Mammalian (Pico v2)는 70 - 400 nt 내의 cDNA를 포함하는 모든 체액으로부터 strand-specific하게 염기서열을 분석할 수 있도록 고안되었으며, 짧은 길이의 cDNA를 포함하는 혈장과 EV 샘플에서는 90 nt 정도에서, 그 외의 샘플에서는 180 - 190 nt 정도에서 피크가 확인되었다. 본 논문의 저자는 각 체액으로부터 ml 당 포함하고 있는 RNA의 양을 상대 정량하였다. ePFP와 cPFP 샘플의 RNA 양은 ePRP, 소변, CM 내 RNA보다 17배 낮았으며, 각 체액 내 EV에서도 CM보다 2,763배, 소변 샘플 보다 7.6배 더 적은 양의 RNA가 있음을 확인하였다. 또한, cPFP와 ePFP에서 추출한 EV에서 유전자 수을 비교했을 때, cPFP EV에서 더 적은 유전자 수를 확인하였다. 이를 통해, Plasma 검체를 채취하는 방법에 따라 유전자 수가 달라질 수 있음을 확인하였고, 실험 적용 시 샘플 채취조건에 대한 최적화가 필요함을 확인하였다.
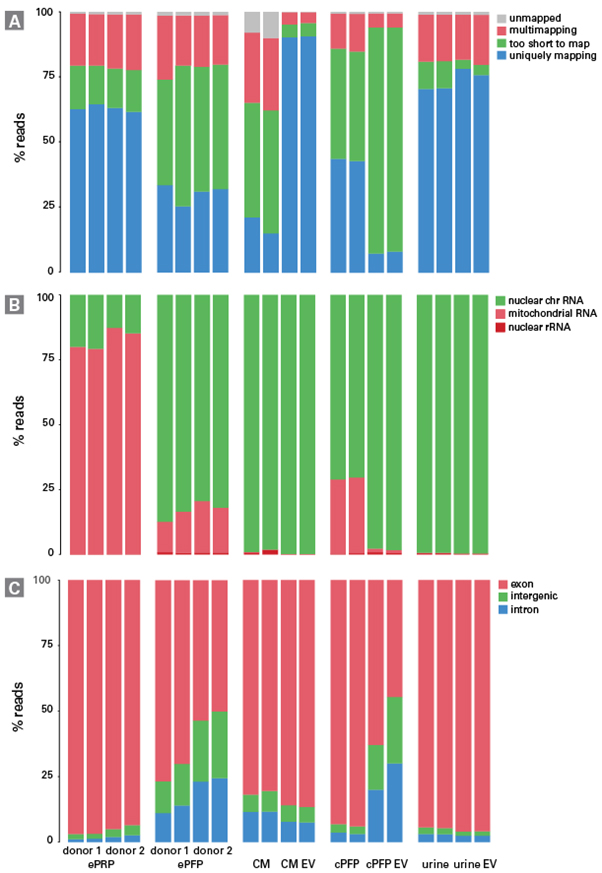
그림 1. 샘플 간 Library 내 read 분포 차이
(Panel A) Percentage of reads assigned as too short to map, uniquely mapping, multi-mapping, or unmapped.
(Panel B) Percentage of reads derived from nuclear RNA, mitochondrial RNA, and ribosomal RNA per sample.
(Panel C) Percentage of the reads originating from nuclear chromosomes derived from exonic, intronic, and intergenic regions per sample. All data was quantified with STAR. Figure used from Everaert et al. 2019 under Creative Commons License 4.0.
[Putting Pico v2 to the test] Reproducibility
SMARTer Stranded Total RNA-Seq 방법은 다양한 인간 체액과 그로부터 추출한 각각의 EV로부터 full-length RNA-seq용 library를 만들기 위해 사용할 수 있으며, 각 체액 샘플 종류에 따라 특이적으로 재현성 높은 분석이 가능한지 확인해 보았다.
ePRP, ePFP 샘플을 이용하여 반복한 후 read counts에 대한 scatter plot을 확인하였을 때, 각각의 샘플로부터 추출한 RNA에서 재현성 있는 결과를 확인하였다 (그림 2).
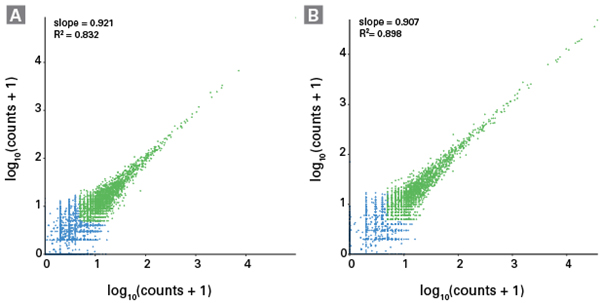
그림 2. ePRP, ePRP에서 분리한 RNA에서 재현성 높은 분석 결과 확인
ePRP (Panel A) and ePFP (Panel B) replicate correlation with filtered (counts <4, blue) and retained genes (counts ≥4, green) resulted in high Pearson correlation of 0.912 and 0.948, respectively. Figure used from Everaert et al. 2019 under Creative Commons License 4.0.
Biological differences in RNA content among biofluids
본 논문의 저자는 인간 체액과 체액에서 추출한 EV를 이용해 RNA 전사체를 분석하여 얻은 유전자 수를 비교해보았다 (표 1). 소변과 소변에서 추출한 EV에서는 굉장히 유사한 유전자가 확인되었으나, 혈장과 혈장에서 추출한 EV에서는 각기 다른 유전자가 확인되었다. 보다 많은 종류의 체액 샘플을 이용함으로써, EV로 인해 운반되거나 운반되지 않는 RNA molecule에 대해 확인할 수 있을 것으로 보인다.
[원문] Be part of the solution: total RNA-seq from human biofluids and extracellular vesicles
[참고문헌]
- Everaert, C. et al. Performance assessment of total RNA sequencing of human biofluids and extracellular vesicles. Sci. Reps. 9, 17574 (2019).
The advantage of the SMARTer® Stranded Total RNA-Seq method is its potential to process low amounts of input material. Indeed, collecting samples is often the bottleneck of fundamental, (pre)clinical and translational research projects and being able to disseminate large amounts of information from only 200 μL (or less) can substantially impact research progress.
The unrealized potential of cell-free RNA
RNA profiling은 사람 체액으로부터 biomarker를 확인하기 위해 사용되는 강력한 도구이다. 액체 생검 분석을 위해 Circulating DNA가 가장 보편적으로 사용되고 있지만, 혈장, 소변, extracellular vesicles (EV) 로부터 추출한 extracellular RNA 또한 질병 상태에 대한 유용한 정보를 제공할 수 있다. 하지만, 체액을 이용해 RNA를 sequencing하는 것은 샘플 양이 적거나, 정량하기 어려울 정도로 RNA가 손상되는 등으로 기술적인 한계가 있었다.
게다가, 최근의 Cell-free 샘플의 total RNA sequencing을 이용한 전사체 분석법은 짧은 fragment, 발현량이 적은 유전자, rRNA의 오염 등으로 인한 분석 한계가 있었다. 이에 대한 대안으로, Ghent 대학의 유전체 연구소와 Biogazelle, Zwijnaarde (Belgium) 연구원들은 다양한 체액과 EV로부터 total RNA를 정확하고, 민감도 있게 정량하기 위해, SMARTer® Stranded Total RNA-Seq Kit - Pico Input Mammalian (Pico v2)로 strand-specific total RNA library를 제작하여 평가하였다.
[Putting Pico v2 to the test] Read distribution and sample type
두 공여자의 platelet-rich plasma in EDTA tube (ePRP), platelet-free plasma in EDTA tube (ePFP)로부터 RNA-seq을 진행하였다. ePRP에서는 75% 이상의 unique reads가 mitochondrial RNA (mtRNA)로부터 유래로 확인된 반면, ePFP는 mtRNA 유래의 reads가 3배 적었고, 많은 reads가 nuclear DNA에 mapping 되었다. 혈장 내 혈소판 유무와 관계없이 unmapped reads와 multi-mapped reads의 수는 비슷하게 확인되었지만, exonic, intronic, intergenic reads 분포는 혈장 내 혈소판 유무에 따라 다르게 나타났다 (그림 1).
함께, 본 논문의 저자는 유방암 세포의 배양 배지 (conditioned medium, CM), 건강한 세 번째 공여자의 platelet-free plasma in citrate tube (cPFP), 전립선 암 환자의 소변 샘플 그리고, 각 샘플 내 EV를 이용하여 염기서열을 분석하였다. cPFP 유래 EV는 7.69%의 unique reads만 확인된 반면 CM 유래 EV에는 90.2%의 unique reads가 확인되었을 만큼 각각의 샘플과 EV에서 다양한 mapping rate를 보였다 (그림 1, Panal A). 거의 대부분의 샘플에서 확인된 Unique reads는 주로 nuclear DNA에 mapping되었으며, 건강한 공여자의 cPFP는 25.8%의 mtRNA를 포함하고 있음을 확인하였다 (그림 2, Panal B). Intron과 intergenic reads를 다수 포함하고 있는 cPFP 유래 EV를 제외하고는, nuclear DNA에 mapping되는 대부분의 reads는 exon 유래임이 확인 되었다 (그림 1, Panal C).
SMARTer® Stranded Total RNA-Seq Kit - Pico Input Mammalian (Pico v2)는 70 - 400 nt 내의 cDNA를 포함하는 모든 체액으로부터 strand-specific하게 염기서열을 분석할 수 있도록 고안되었으며, 짧은 길이의 cDNA를 포함하는 혈장과 EV 샘플에서는 90 nt 정도에서, 그 외의 샘플에서는 180 - 190 nt 정도에서 피크가 확인되었다. 본 논문의 저자는 각 체액으로부터 ml 당 포함하고 있는 RNA의 양을 상대 정량하였다. ePFP와 cPFP 샘플의 RNA 양은 ePRP, 소변, CM 내 RNA보다 17배 낮았으며, 각 체액 내 EV에서도 CM보다 2,763배, 소변 샘플 보다 7.6배 더 적은 양의 RNA가 있음을 확인하였다. 또한, cPFP와 ePFP에서 추출한 EV에서 유전자 수을 비교했을 때, cPFP EV에서 더 적은 유전자 수를 확인하였다. 이를 통해, Plasma 검체를 채취하는 방법에 따라 유전자 수가 달라질 수 있음을 확인하였고, 실험 적용 시 샘플 채취조건에 대한 최적화가 필요함을 확인하였다.

그림 1. 샘플 간 Library 내 read 분포 차이
(Panel A) Percentage of reads assigned as too short to map, uniquely mapping, multi-mapping, or unmapped.
(Panel B) Percentage of reads derived from nuclear RNA, mitochondrial RNA, and ribosomal RNA per sample.
(Panel C) Percentage of the reads originating from nuclear chromosomes derived from exonic, intronic, and intergenic regions per sample. All data was quantified with STAR. Figure used from Everaert et al. 2019 under Creative Commons License 4.0.
[Putting Pico v2 to the test] Reproducibility
SMARTer Stranded Total RNA-Seq 방법은 다양한 인간 체액과 그로부터 추출한 각각의 EV로부터 full-length RNA-seq용 library를 만들기 위해 사용할 수 있으며, 각 체액 샘플 종류에 따라 특이적으로 재현성 높은 분석이 가능한지 확인해 보았다.
ePRP, ePFP 샘플을 이용하여 반복한 후 read counts에 대한 scatter plot을 확인하였을 때, 각각의 샘플로부터 추출한 RNA에서 재현성 있는 결과를 확인하였다 (그림 2).

그림 2. ePRP, ePRP에서 분리한 RNA에서 재현성 높은 분석 결과 확인
ePRP (Panel A) and ePFP (Panel B) replicate correlation with filtered (counts <4, blue) and retained genes (counts ≥4, green) resulted in high Pearson correlation of 0.912 and 0.948, respectively. Figure used from Everaert et al. 2019 under Creative Commons License 4.0.
Biological differences in RNA content among biofluids
본 논문의 저자는 인간 체액과 체액에서 추출한 EV를 이용해 RNA 전사체를 분석하여 얻은 유전자 수를 비교해보았다 (표 1). 소변과 소변에서 추출한 EV에서는 굉장히 유사한 유전자가 확인되었으나, 혈장과 혈장에서 추출한 EV에서는 각기 다른 유전자가 확인되었다. 보다 많은 종류의 체액 샘플을 이용함으로써, EV로 인해 운반되거나 운반되지 않는 RNA molecule에 대해 확인할 수 있을 것으로 보인다.
Sample |
Shared genes |
Unique genes |
Unique to EV |
Conditioned medium |
4,891 |
755 |
1,853 |
Urine |
>10,000 |
521 |
900 |
cPFP |
1,528 |
7,211 |
70 |
[원문] Be part of the solution: total RNA-seq from human biofluids and extracellular vesicles
[참고문헌]
- Everaert, C. et al. Performance assessment of total RNA sequencing of human biofluids and extracellular vesicles. Sci. Reps. 9, 17574 (2019).